Nuovo metodo per mappare le proteine
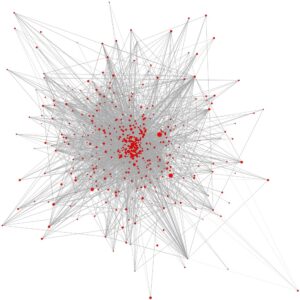
Mappa interazione Proteine umane
Dopo la mappatura del genoma umano, la nuova sfida e’ quella di mappare le proteine. Il compito e’ molto piu’ arduo, perche’ mentre i geni sono poco piu’ di 20 mila, le molecole proteiche che compongono il corpo umano sono milioni.
“Stiamo cambiando radicalmente la strategia per la comprensione delle molecole proteiche, e’ come essere alla vigilia del progetto Human Proteome”. “Abbiamo bisogno di definire tutte le molecole di proteine nel corpo umano”, ha detto Kelleher. “In primo luogo – ha continuato – abbiamo bisogno di una mappa di forme di proteine sane, che servira’ da riferimento per comprendere le forme proteiche danneggiate e malate. La nostra tecnologia ci permettera’ di percorrere velocemente questa strada”. La squadra di Kelleher ha sviluppato un sistema a quattro dimensioni che utilizza la spettrometria di massa per misurare la massa di carica e il peso di ogni proteina.
Partendo da questi elementi, il software sviluppato dal team riuscira’ a effettuare una ricostruzione top-down delle molecole proteiche. Nella prima dimostrazione su larga scala del metodo top-down, i ricercatori sono stati in grado di identificare oltre 3.000 forme di proteine create da 1.043 geni. “Siamo alla vigilia di un grande inizio”, ha concluso il ricercatore.