Scienziati scoprono meccanismo di regolazione del gene umano
Scienziati provenienti da Germania, Spagna e Francia affermano che, se si vogliono produrre proteine, bisogna trascrivere un gene che codifica le proteine nell’acido ribonucleico (RNA) e nel “processo di splicing” ridotto in modo corretto.
Nel loro ultimo studio, presentato nella rivista Nature, i ricercatori forniscono nuove informazioni sul modo in cui la proteina U2AF rende possibile questo processo. La ricerca è stata in parte finanziata da tre progetti UE con un totale di 25,4 milioni di euro nell’ambito del Sesto programma quadro (6°PQ).
Guidati dal Helmholtz Zentrum München e dal Politecnico di Monaco (TUM) in Germania, i ricercatori hanno scoperto come la proteina U2AF attiva l’RNA messaggero precursore (pre-mRNA) che serve da modello per la sintesi proteica nel corpo. Il pre-mRNA deve dapprima essere trascritto dall’acido deossiribonucleico (DNA).
Varie proteine, ciò che gli esperti chiamano i fattori di splicing, devono lavorare assieme per garantire il successo dello splicing, che gioca un ruolo cruciale nel sistema centrale della biologia molecolare. Vale a dire che i dati genetici si muovono in una direzione: dal DNA all’RNA, e quindi alle proteine.
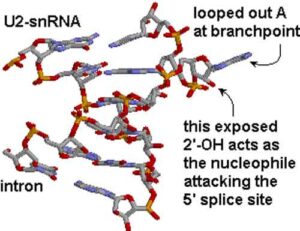
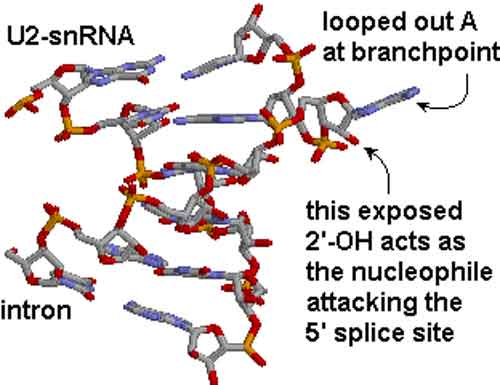
Vi è una struttura specifica contenuta nei geni del genoma umano. Le aree con gli esoni chiave si alternano ad aree che i ricercatori chiamano introni, che contengono dati irrilevanti che non codificano la proteina corrispondente.
Alcuni membri del team hanno valutato la U2AF; essa è un fattore di splicing formato da due moduli strutturali che si lega all’RNA in prossimità del limite tra introne ed esone. La copia del pre-mRNA subisce lo splicing con la rimozione degli introni. In questo modo il mRNA rimanente è composto soltanto da esoni, che codificano la sequenza di amminoacidi di una data proteina.
“La struttura spaziale della proteina U2AF alterna tra una conformazione chiusa e una aperta,” spiega il professor Michael Sattler, direttore dell’Istituto di biologia strutturale presso l’Helmholtz Zentrum München e professore di Spettroscopia biomolecolare NMR [risonanza magnetica nucleare] al TUM. “Una sequenza RNA corrispondente nell’introne fa sì che la U2AF assuma una conformazione aperta che attiva lo splicing e alla fine porta alla rimozione dell’introne.”
Secondo i ricercatori, la sequenza RNA dell’introne determina con quanta efficacia possa essere attivato questo cambio conformazionale. Un processo di selezione conformazionale aiuta a provocare un cambio di equilibrio tra la forma chiusa e quella aperta della proteina U2AF. In effetti, l’RNA si lega a una minuscola frazione della conformazione aperta esistente, indipendentemente dalla presenza o dall’assenza di RNA. Essi ritengono che meccanismi simili possano contribuire in modo significativo alla regolazione di un alto numero di altre vie di trasduzione del segnale nella cellula.
“I nostri risultati indicano che i domini RRM in tandem di U2AF65 non agiscono semplicemente da supporto per il legame, ma hanno invece un ruolo attivo nel mettere in relazione in modo quantitativo la forza del tratto Py al riconoscimento del sito di splicing e all’assemblaggio dello spliceosoma,” scrivono gli autori. “La selezione conformazionale multidominio degli stati aperti permette ai domini RRM in tandem di funzionare da reostato molecolare per l’attività della U2AF durante le prime fasi dello splicing, che vedono una competizione per il RRM1 tra il RRM2 legante (auto inibizione in una conformazione chiusa) e l’RNA (attivazione mediante una conformazione aperta). Ciò fornisce un filtro di selettività contro il legame libero dell’RNA e l’assemblaggio dello spliceosoma, poiché i ligandi a più alta affinità del tratto Py sono meglio in grado di annullare il costo energetico necessario per il legame di entrambi i domini RRM.”
Lo studio è stato finanziato dai seguenti progetti UE, tutti nell’ambito dell’area tematica “Scienze della vita, genomica e biotecnologie per la salute” del 6°PQ: 3D-REPERTOIRE, FSG-V-RNA, ed EURASNET.
3D-REPERTOIRE (“A multidisciplinary approach to determine the structures of protein complexes in a model organism”) ha ricevuto finanziamenti pari a 13 milioni di euro. FSG-V-RNA (“Functional and structural genomics of viral RNA”) è stato supportato con 2,4 milioni di euro, mentre EURASNET (“European alternative splicing network of excellence”) ha ricevuto 10 milioni di euro.
Per maggiori informazioni, visitare:
Nature:
http://www.nature.com/
Helmholtz Zentrum München:
http://www.helmholtz-muenchen.de/en/start/index.html